利用ggseqlogo绘制seqlogo图
简介sequence logo图用来可视化一段序列某个位点的保守性,据根提供的序列组展示位点信息。这方面有很多在线小工具可以完成,这里使用R包ggseqlogo进行可视化。
安装安装方式有两种#直接从CRAN中安装install.packages(“ggseqlogo”)#从GitHub中安装devtools::install.github(“omarwagih/ggseqlogo”)
数据加载ggseqlogo提供了测试数据ggseqlogo_sample。
#加载包library(ggplot2)library(ggseqlogo)
#加载数据data(ggseqlogo_sample)ggseqlogo_sample数据集是一个列表,里面包含了三个数据集:seqs_dna:12种转录因子的结合位点序列pfms_dna:四种转录因子的位置频率矩阵seqs_aa:一组激动酶底物磷酸化位点序列#seqs_dnahead(seqs_dna)[1]
## $MA0001.1## [1] “CCATATATAG” “CCATATATAG” “CCATAAATAG” “CCATAAATAG” “CCATAAATAG”## [6] “CCATAAATAG” “CCATAAATAG” “CCATATATGG” “CCATATATGG” “CCAAATATAG’
#pfms_dnahead(pfms_dna)[1]
## $MA0018.2## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8]## A 0 0 11 0 1 0 2 8## C 1 1 0 9 0 3 7 0## G 1 10 0 2 10 0 1 1## T 9 0 0 0 0 8 1 2
#seqs_aahead(seqs_aa)[1]
## $AKT1## [1] “VVGARRSSWRVVSSI” “GPRSRSRSRDRRRKE” “LLCLRRSSLKAYGNG”## [4] “TERPRPNTFIIRCLQ” “LSRERVFSEDRARFY” “PSTSRRFSPPSSSLQ”
可视化ggplot()+geom_logo(seqs_dna$MA0001.1)+theme_logo()

ggseqlogo提供了一个直接绘图的函数ggseqlogo(),这是一个包装函数。下面命令结果同上面的。
ggseqlogo(seqs_dna$MA0001.1)
输入格式ggseqlogo支持以下几种类型数据输入:序列矩阵下面是使用数据中的位置频率矩阵生成的seqlogoggseqlogo(pfms_dna$MA0018.2)

方法ggseqlogo通过method选项支持两种序列标志生成方法:bits和probability。p1 <- ggseqlogo(seqs_dna$MA0001.1, method=”bits”)p2 <- ggseqlogo(seqs_dna$MA0001.1, method=”prob”)gridExtra::grid.arrange(p1,p2)
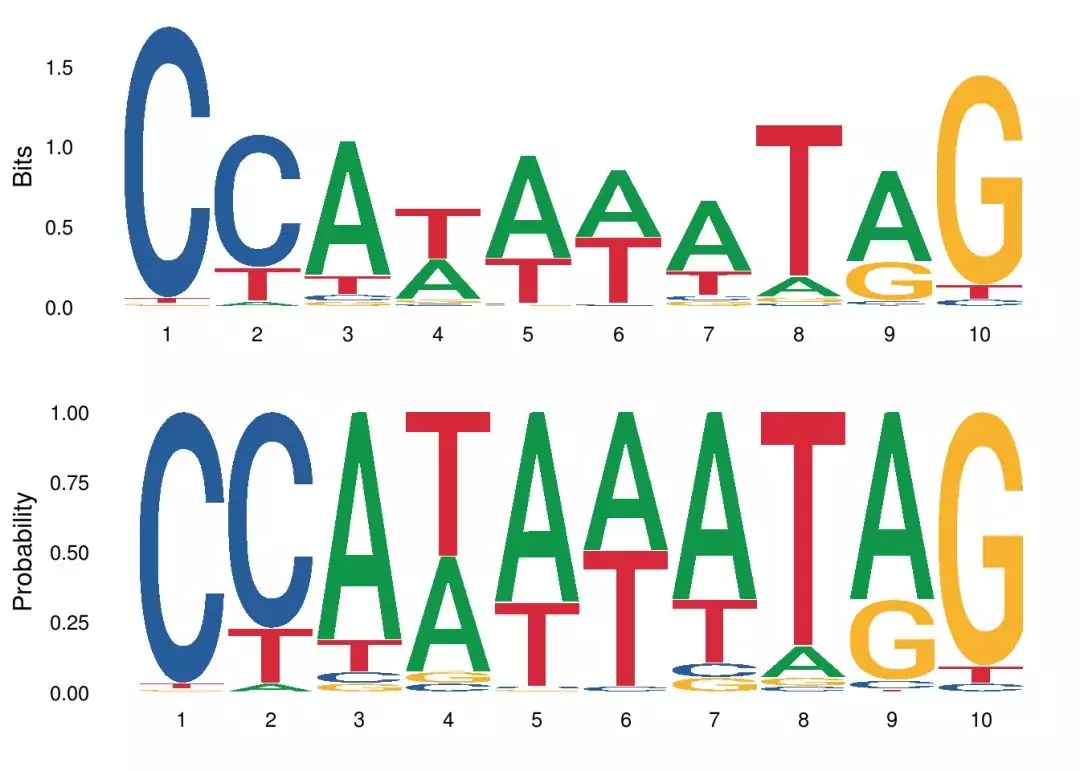
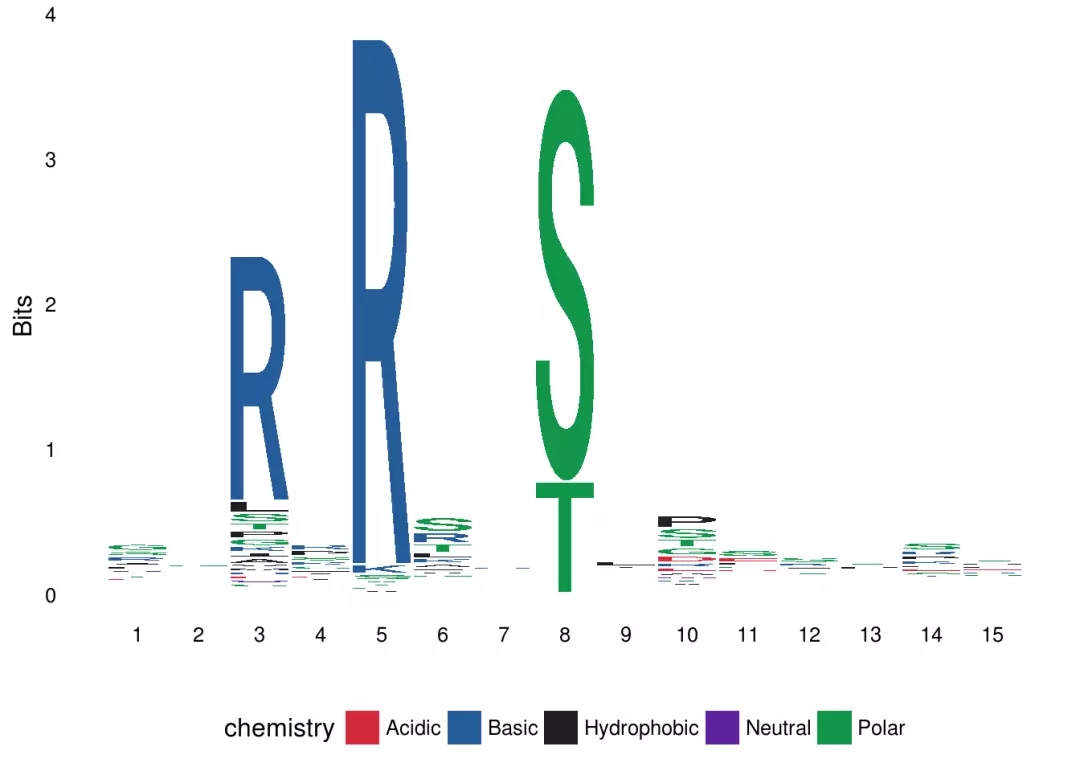
序列类型ggseqlogo支持氨基酸、DNA和RNA序列类型,默认情况下ggseqlogo会自动识别数据提供的序列类型,也可以通过seq_type选项直接指定序列类型。ggseqlogo(seqs_aa$AKT1, seq_type=”aa”)
自定义字母通过namespace选项来定义自己想要的字母类型#用数字来代替碱基seqs_numeric <- chartr(“ATGC”, “1234”, seqs_dna$MA0001.1)ggseqlogo(seqs_numeric, method=”prob”, namespace=1:4)

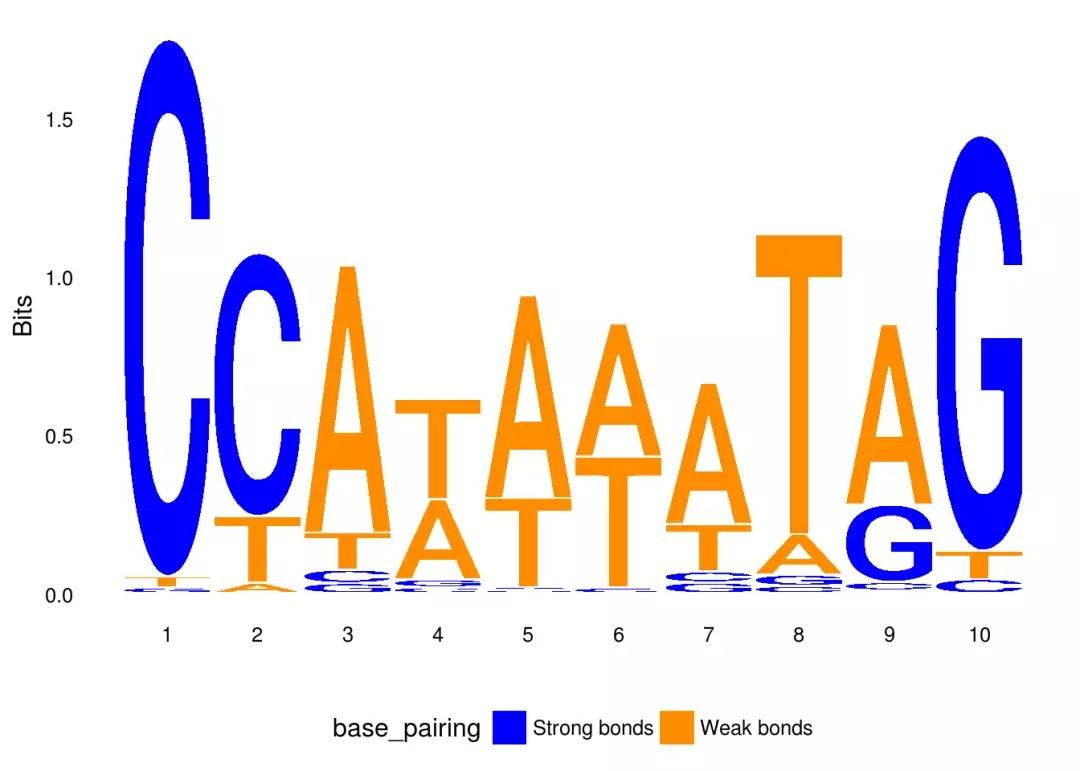
配色ggseqlogo可以使用col_scheme参数来设置配色方案,具体可参考?list_col_schemesggseqlogo(seqs_dna$MA0001.1, col_scheme=”base_pairing”)
自定义配色ggseqlogo提供函数make_col_scheme来自定义离散或者连续配色方案离散配色”)csl <- make_col_scheme(chars = c(“A”,”T”, “C”, “G”), groups = c(“gr1″,”gr1”, “gr2″,”gr2”), cols = c(“purple”,”purple”,”blue”,”blue”))ggseqlogo(seqs_dna$MA0001.1,col_scheme=csl)

连续配色cs2 <- make_col_scheme(chars = c(“A”, “T”, “C”, “G”), values = 1:4)ggseqlogo(seqs_dna$MA0001.1, col_scheme=cs2)

同时绘制多个序列标志ggseqlogo(seqs_dna, ncol = 4)
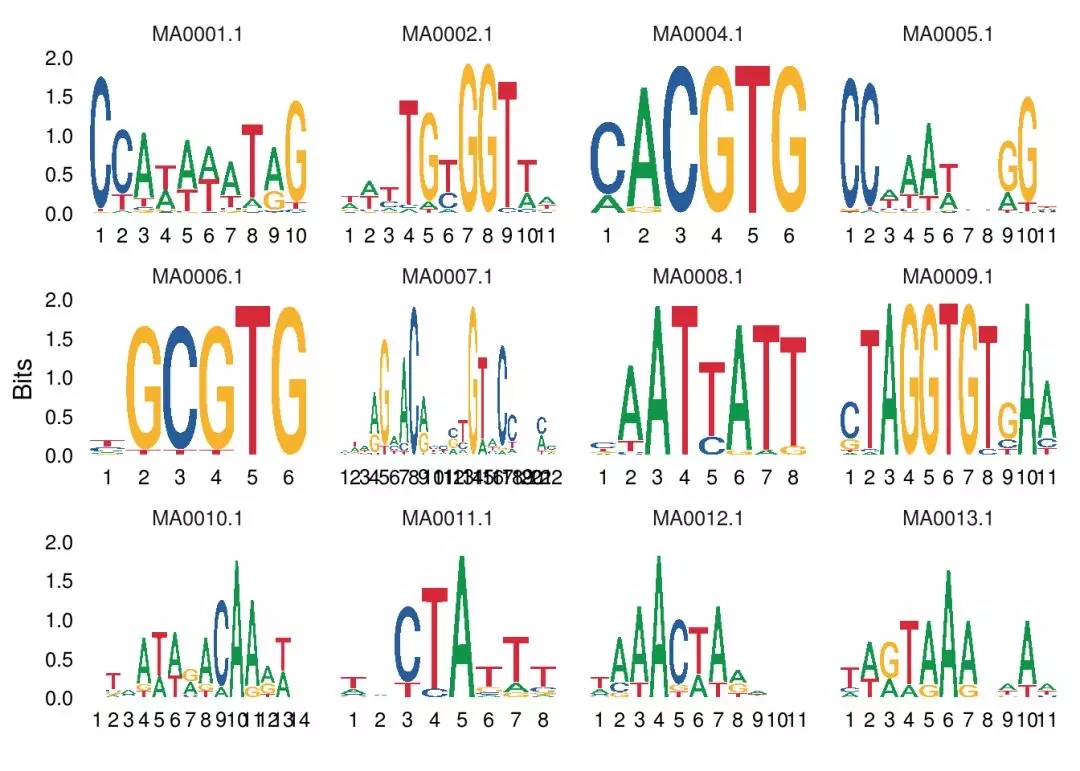
上述命令实际上等同于ggplot()+geom_logo(seqs_dna)+theme_logo()+facet_wrap(~seq_group,ncol = 4,scales = “free_x”)
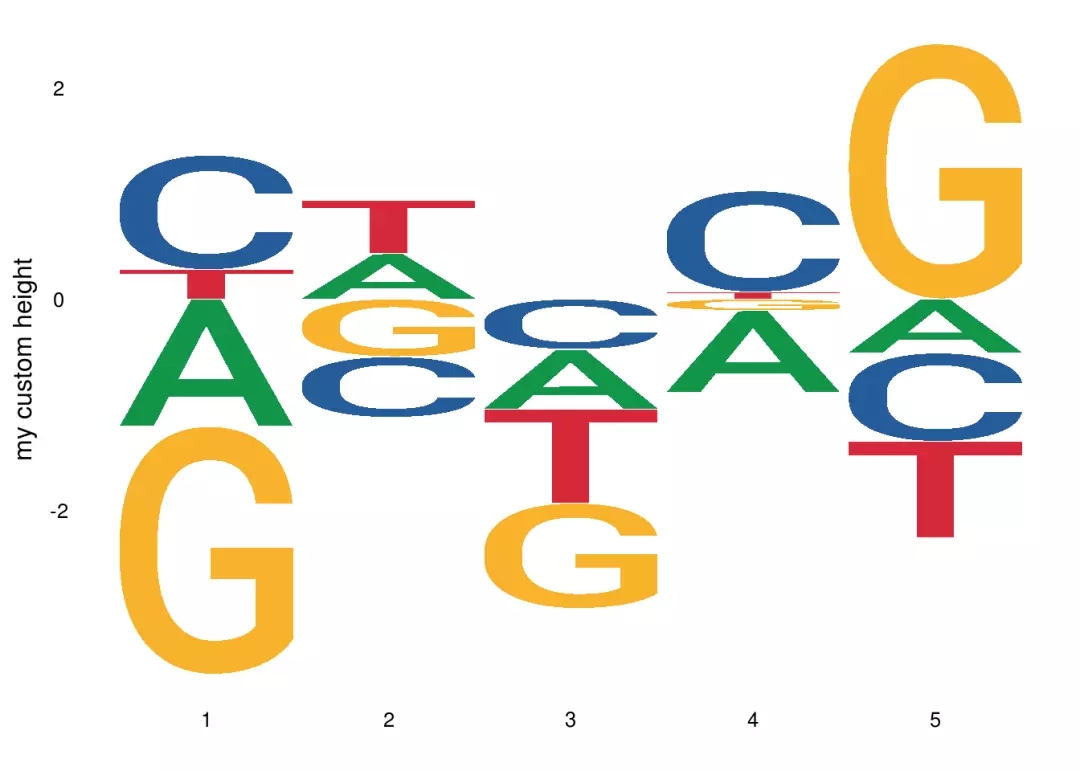
自定义高度通过创建矩阵可以生成每个标志的高度,还可以有负值高度set.seed(1234)custom_mat <- matrix(rnorm(20), nrow = 4, dimnames = list(c(“A”,”T”,”C”, “G”)))ggseqlogo(custom_mat,method=”custom”,seq_type=”dna”)+ylab(“my custom height”)
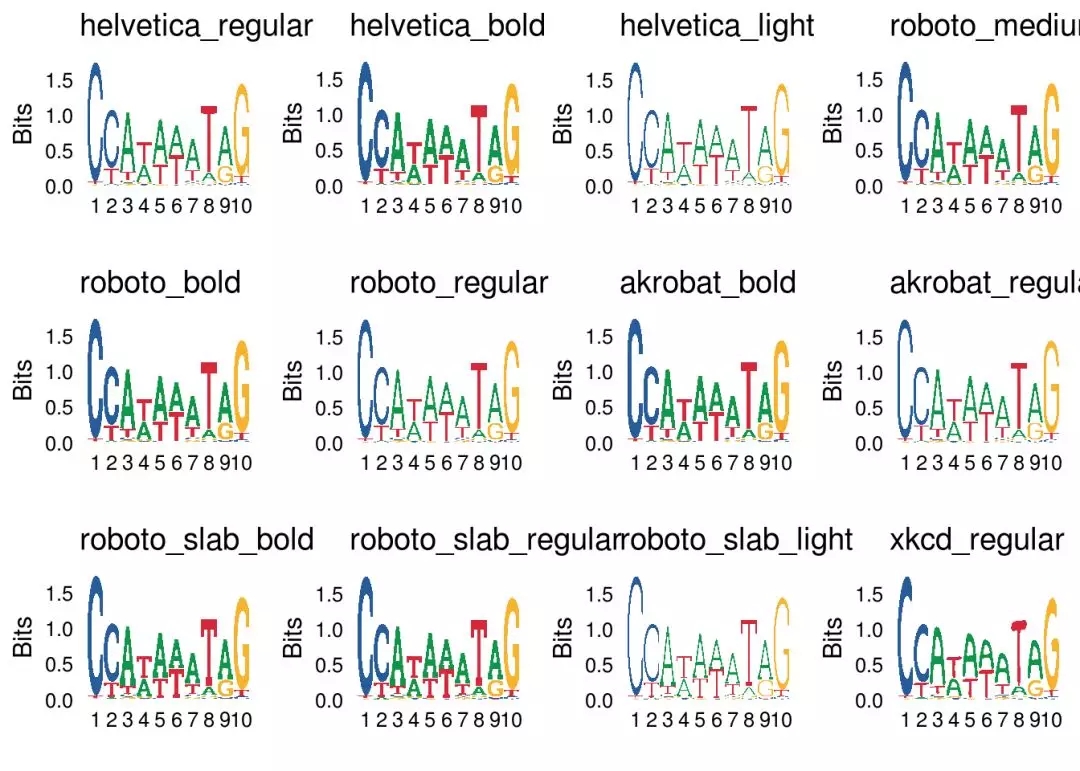
字体可以通过font参数来设置字体,具体可参考?list_fontsfonts <- list_fonts(F)p_list <- lapply(fonts, function(f){ggseqlogo(seqs_dna$MA0001.1,font=f)+ggtitle(f)})do.call(gridExtra::grid.arrange,c(p_list, ncol=4))
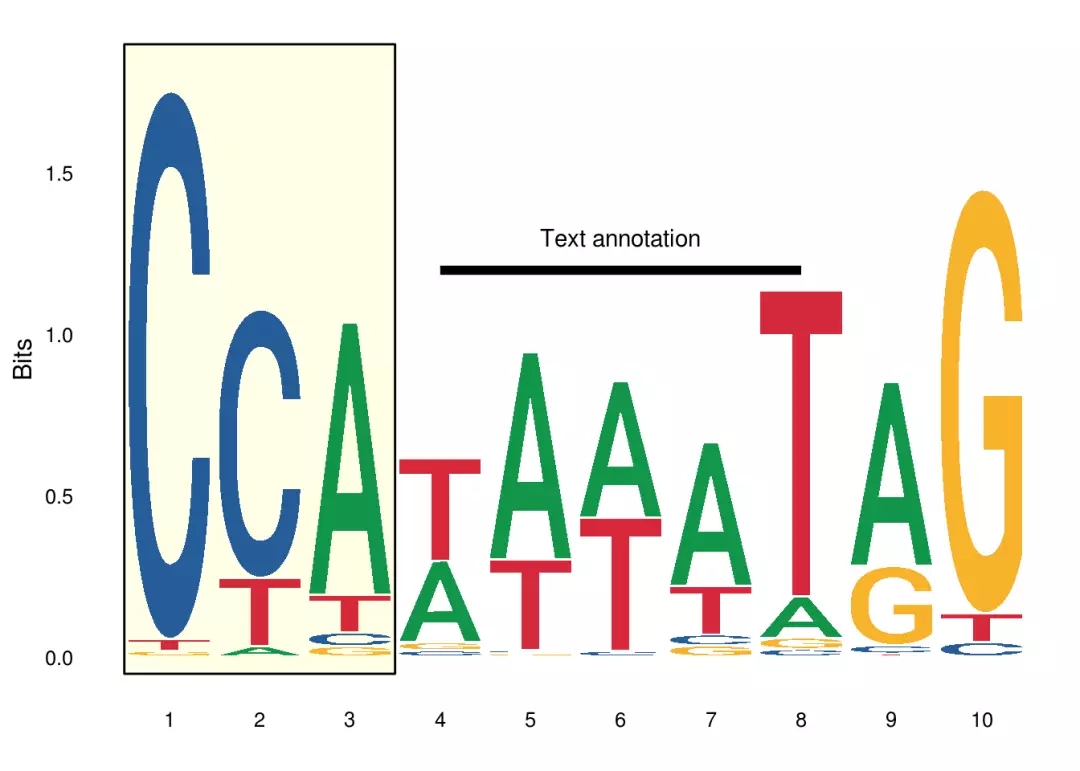
注释注释的话跟ggplot2是一样的ggplot()+annotate(“rect”, xmin = 0.5, xmax = 3.5, ymin = -0.05, ymax = 1.9, alpha=0.1, col=”black”, fill=”yellow”)+geom_logo(seqs_dna$MA0001.1, stack_width = 0.9)+annotate(“segment”, x=4, xend = 8, y=1.2, yend = 1.2, size=2)+annotate(“text”, x=6, y=1.3, label=”Text annotation”)+theme_logo()
图形组合将ggseqlogo生成的图形与ggplot2生成的图形组合在一起。p1 <- ggseqlogo(seqs_dna$MA0008.1)+theme(axis.text.x = element_blank())aln <- data.frame(letter=strsplit(“AGATAAGATGATAAAAAGATAAGA”, “”)[[1]],species=rep(c(“a”,”b”,”c”), each=8),x=rep(1:8,3))aln$mut <- “no”aln$mut[c(2,15,20,23)]=”yes”p2 <- ggplot(aln, aes(x, species)) +geom_text(aes(label=letter, color=mut, size=mut)) +scale_x_continuous(breaks=1:10, expand = c(0.105, 0)) + xlab(”) +scale_color_manual(values=c(‘black’, ‘red’)) +scale_size_manual(values=c(5, 6)) +theme_logo() +theme(legend.position = ‘none’, axis.text.x = element_blank())bp_data <- data.frame(x=1:8,conservation=sample(1:100, 8))p3 <- ggplot(bp_data, aes(x, conservation))+geom_bar(stat = “identity”, fill=”grey”)+theme_logo()+scale_x_continuous(breaks = 1:10, expand = c(0.105, 0))+xlab(“”)suppressMessages(require(cowplot))plot_grid(p1,p2,p3,ncol = 1, align = “v”)
#p#分页标题#e#

SessionInfo## R version 3.4.3 (2017-11-30)## Platform: x86_64-pc-Linux-gnu (64-bit)## Running under: Ubuntu 17.10#### Matrix products: default## BLAS: /usr/lib/x86_64-linux-gnu/atlas/libblas.so.3.10.3## LAPACK: /usr/lib/x86_64-linux-gnu/atlas/liblapack.so.3.10.3#### locale:## [1] LC_CTYPE=zh_CN.UTF-8 LC_NUMERIC=C## [3] LC_TIME=zh_CN.UTF-8 LC_COLLATE=zh_CN.UTF-8## [5] LC_MONETARY=zh_CN.UTF-8 LC_MESSAGES=zh_CN.UTF-8## [7] LC_PAPER=zh_CN.UTF-8 LC_NAME=C## [9] LC_ADDRESS=C LC_TELEPHONE=C## [11] LC_MEASUREMENT=zh_CN.UTF-8 LC_IDENTIFICATION=C#### attached base packages:## [1] stats graphics grDevices utils datasets methods base#### other attached packages:## [1] cowplot_0.9.2 ggseqlogo_0.1 ggplot2_2.2.1#### loaded via a namespace (and not attached):## [1] Rcpp_0.12.15 knitr_1.20 magrittr_1.5 munsell_0.4.3## [5] colorspace_1.3-2 rlang_0.2.0 stringr_1.3.0 plyr_1.8.4## [9] tools_3.4.3 grid_3.4.3 gtable_0.2.0 htmltools_0.3.6## [13] yaml_2.1.16 lazyeval_0.2.1 rprojroot_1.3-2 digest_0.6.15## [17] tibble_1.4.2 gridExtra_2.3 evaluate_0.10.1 rmarkdown_1.8## [21] labeling_0.3 stringi_1.1.6 compiler_3.4.3 pillar_1.1.0## [25] scales_0.5.0 backports_1.1.2
欢迎加入本站公开兴趣群商业智能与数据分析群兴趣范围包括各种让数据产生价值的办法,实际应用案例分享与讨论,分析工具,ETL工具,数据仓库,数据挖掘工具,报表系统等全方位知识QQ群:81035754
