操作R的glm函数举办卵白质“热点”氨基酸的模子预测
生物信息学是一门较量新兴学科,并且保罗万象。主要的事情会合于对DNA序列的数据,卵白质氨基酸序列信息举办阐明事情(包罗软件开拓,算法创新等)而对卵白质三维布局的信息的阐明也是个中之一。
由于卵白质的三维布局简直定需要比DNA序列测序,卵白质序列测序更为巨大的尝试完成,因此有关数据会越发稀少,而且各人也都在摸索如何通过测定个中的一些空间布局参数,可以或许实现较准确的预测程度,淘汰尝试耗损,提高方针性。
而本题目阐明的hotspot,即卵白质上的“热点”指的是在卵白质与卵白质彼此之间产生彼此浸染而起到生理浸染的时候,在打仗面上相对更为重要的氨基酸。也就是两者外貌大概会有化学键的形成而不变布局等的成果。而是否重要的一个权衡指标就是通过尝试技能改变卵白质某打仗外貌的一个氨基酸而改换为一个在空间上根基没有什么孝敬的丙氨酸,而且丈量改换之后G(吉布斯自由能)的变革环境。假如变革越大证明本来的氨基酸在团结中的浸染更大,因为普遍意义上的卵白质的最不变状态就是低落自身的能量状态,淘汰化学回响大概造成的成果失活。
但就像上面所说,通过尝试来举办测定的话,则整体的事情量将会很大,因为你要一个一个举办替换而且测定能量变革。可是假如尝试前有预先预期的大概的hotspot的位点话,则能加速尝试的历程,提高效率,而且为卵白质的改革和从头设计等都有指导意义。因此,已经有许多的人对此刻已经有的尝试功效举办阐明,试图探寻更好的预测参数和更好的模子举办预测。而这里的数据来自一篇关于新的算法的文章的附录数据集。(文章地点http://bioinformatics.oxfordjournals.org/content/25/12/1513.abstract)而个中的数据也是来自网上别人通过文献收集获得的一个相关数据库中得到,别离为Alanine Scanning Energetics Database (ASEdb).和Binding Interface Database (BID)
个中的各行参数意义(第一行为打仗面,个中的编号如1a4yAB代表在一个叫做PDB的3D卵白布局数据库中http://www.rcsb.org/pdb/home/home.do的卵白编号和卵白的浸染肽链的编号(此处既是1a4y号卵白,个中的A和B两条链在空间中的打仗面)
第二行为氨基酸残基的编号,20个氨基酸每个有一个大写字母暗示,如W261暗示色氨酸,位于A链条的第261个位置。)
尔后头的数据包罗各类的回响空间信息的指标,详细可以参考文章中的详细计较进程。而ΔΔGobs等于尝试测得的吉布斯能量变革,默认在变革>1时即为hotspot。而Robetta为一个参照算法,为利用较为遍及的预测软件。其指标也是当>1时即认定为hotspot。
以下流程纯粹为凭据教科书式的模子设计调解,详细可拜见薛毅《统计建模与R软件》6.6广义线性回归模子,出格是例6.20……
>hotspot=read.csv(“hotspot.csv”) #读取数据
>glm1=glm(Observed~Robetta+CompASA+Potential+DiffASA,family=binomial,data=hotspot)
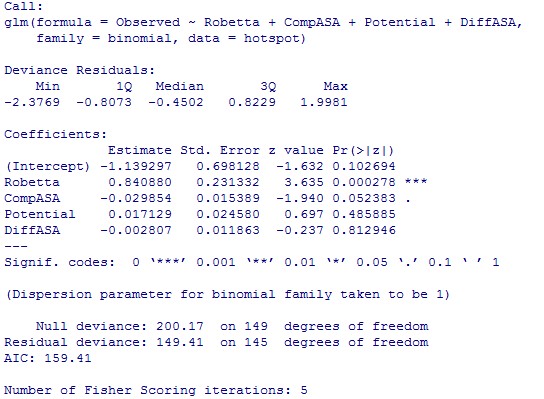
> summary(glm1) #数据全添上再说!
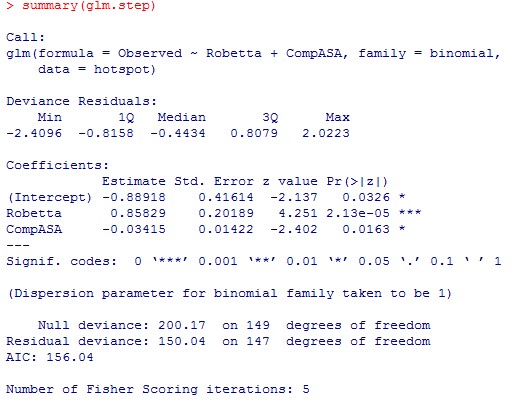
> glm.step=step(glm1) #进一步模子优化(自动……)
> pre=predict(glm.step,data=hotspot)
> p=exp(pre)/(1+exp(pre));p
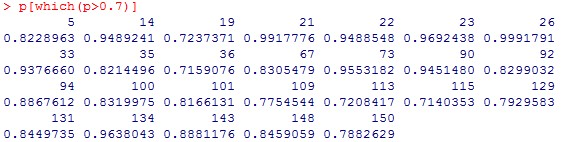
> p[which(p>0.7)] #相应的预测概率计较
> a=which(hotspot$Observed==1)#原先所知道的hotspot为第几号数据
> a
[1] 5 6 7 14 17 18 19 21 22 23 24 25 26 29 30 31 33 34
[19] 35 36 39 50 52 65 67 68 69 70 71 72 73 74 75 76 77 91
[37] 92 94 97 99100 101 102 105 108 109 110 111 113 115 127 129 130 131
[55] 134 135 140 143
hotspot$X=c(1:150)
> b=hotspot$X[which(p>0.6)] #设定概率大于必然值时即认为大概为hotspot
> length(b)
[1] 31
> length(a)
[1] 58
> intersect(a,b)
[1] 5 7 14 19 21 22 23 24 26 33 35 36 67 73 76 92 94 100
[19] 101 108 109 113 115 129 131 134 143
> length(intersect(a,b))
[1] 27
可以看到,在一共58个阳性的环境下,可以或许预测出27个为正确。而31概率大于0.6的中只有4个为错误预测。因此总体来说,模子还过得去咯!?
接下来可以用这个模子做新数据的预测等等…………此处临时就不演示了……
恩,接待各人举办无情的批斗和改造,感谢!~(感激布丁同志啦……)
 dataset.zip
dataset.zip