深度学习命名实体识别第一篇:注释方法公共和数据集
欢迎来的我的博客,这是我的第一篇关于深度学习的文章
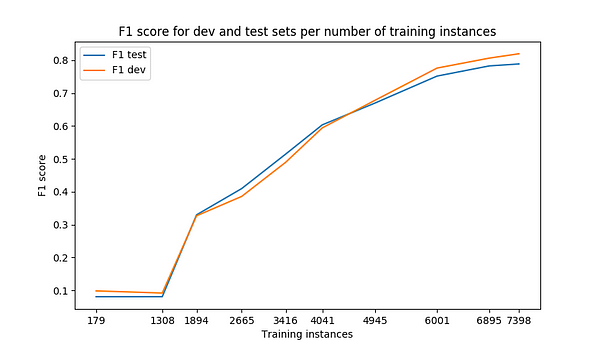
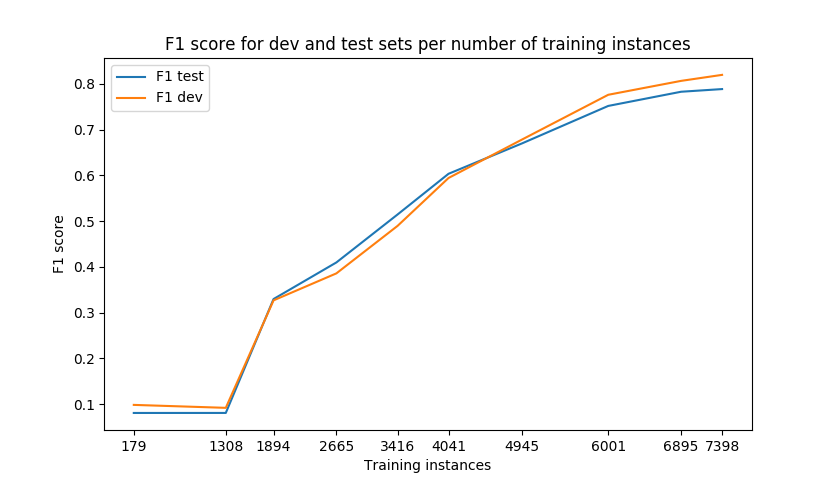
需要数据:NER的深度学习需要数千个训练点才能达到合理的准确性。
在写关于使用深度学习进行命名实体识别(NER)的硕士论文时,我将通过一系列帖子分享我的学习内容。主题包括如何以及在哪里找到有用的数据集(本文!),最新的实现以及今年末一系列关于深度学习模型的优缺点。
公开数据集
与任何深度学习模型一样,您需要一个TON数据。高质量的数据集是任何深度学习项目的基础。幸运的是,在那里有几个注释的,公开可用的和大多数免费的数据集。
CoNLL 2003
这个数据集:https://www.clips.uantwerpen.be/conll2003/ner/包括1,393个英文和909个德文新闻文章。英语语料库是免费的,但不幸的是德国语料库的价格为75美元。这是在这篇文章中花费一些东西的唯一语料库。要构建英文语料库,您需要RCV1 Reuters:https://trec.nist.gov/data/reuters/reuters.html语料库。您将在提交组织和个人协议后几天获得访问权限,且不收取任何费用。
实体用LOC(位置),ORG(组织),PER(人)和MISC(杂项)注释。这是一个例句,每行由[word] [POS tag] [chunk tag] [NER tag]组成:
UN NNP I-NP I-ORG
官方 NN I-NP O
Ekeus NNP I-NP I-PER
头 VBZ I-VP O
用于 IN I-PP O
巴格达 NNP I-NP I-LOC
。 。OO
这里有一个全面的POS标签代表的名单。以下是官方的CoNLL 2003介绍文章和一个带SOTA排名的GitHub wiki。
OntoNotes 5.0 / CoNLL 2012
OntoNotes Release 5.0由来自一系列来源的1,745 K英语,900 K中文和300 K阿拉伯语文本数据组成:电话交谈,新闻通讯,广播新闻,广播对话和网络博客。实体被标注为PERSON,组织和位置类等(18类完整列表在这里,第21页)。通过您的大学/部门访问是最容易的 – 请检查您在此注册的时间。
i2b2挑战
整合生物学和床边的信息学(i2b2)中心已经发布了NER 的一些临床数据集。特别是2009年(提取药物),2012年(提取问题,治疗等)和2014年(提取疾病,风险因素,药物等)挑战是非常相关的,并包括完备的最先进的实施方案。获取访问是免费的 – 它需要签署一份协议,声明你基本上会考虑数据。i2b2很快回复!
更多数据
我没有详细查看其他数据集,但它们可能对您的应用仍然有用:使用蛋白质/ DNA / RNA /细胞系/细胞类型(2,404 MEDLINE摘要)标记的NLPBA 2004和使用名称/日期/时间标记的Enron电子邮件(〜500 K消息)。
注释方法
围绕不同的注释模式开发可能需要一些时间。从标记方法(类HTML)到键值对,有许多框架。
标记(例如,OntoNotes 5.0)
此批注方法使用标记标签使用尖括号来定义命名实体,例如组织:
<ENAMEX TYPE =“ORG”>迪士尼</ ENAMEX>是一个全球品牌。
IOB(例如CoNLL 2003)
IOB(或BIO)代表B egin,I nside和O utside。用O标记的单词不在命名实体之外,而I-XXX标记用于类型为XXX的命名实体内的单词。只要两个类型为XXX的实体彼此紧邻,第二个实体的第一个单词将被标记为B-XXX,以突出显示它启动另一个实体。这里是一个例句,每行由[word] [POS tag] [NER tag]组成:
我们 PRP B-NP
看到 VBDØ
的 DT B-NP
黄 JJ I-NP
狗 NN I-NP
BIOES
更复杂的注释方法区分命名实体的末端和单个实体。这种方法称为BIOES,用于B egin ,I nside ,O utside,E nd,S ingle。以上数据集均不使用BIOES,但与BIO相比,其性能有显着提高(如Chiu和Nichols,2016)。
更多方法和细节
这里有一篇很棒的博客文章,讲述他们复杂的进一步注释方法。
第一篇文章结束。了解可用的数据以及注释的方式为您以后构建可靠的统计模型奠定了良好的基础。让我知道您是否找到其他有用的数据源!
翻译:天才写手
ref:https://towardsdatascience.com/deep-learning-for-ner-1-public-datasets-and-annotation-methods-8b1ad5e98caf
